R绘图 | 如何改变circos图中不同track中sector间的比例
实际案例及问题
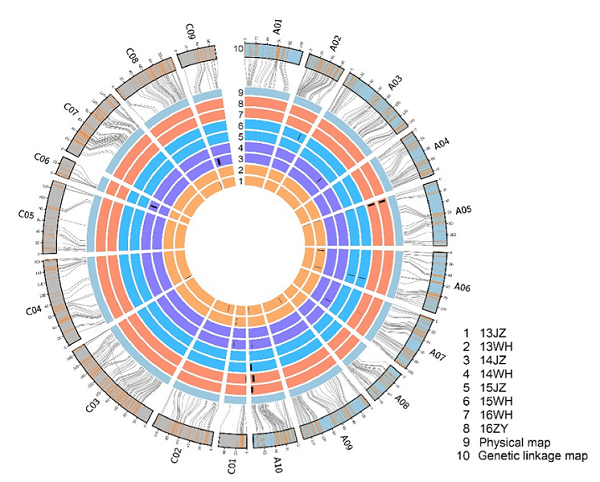
我们先看文章中的例子。在下图中最外两层分别表示了遗传连锁图和物理图(基因组),中间连线代表构图标记在遗传图和物理图上位置的对应关系。我们可以看到不同染色体间的长度比例和不同连锁图间的长度比例是相同的,但是这一点显然和实际情况不符。这幅图是由circos软件绘制,造成上述比例问题的原因可能是circos并不能改变不同track的比例,或者有改变比例的设置方法但作者并不知道。
解决方案
我们今天就尝试着用R语言的ggplot2来解决这一问题。基本思路是先用geom_rect()构建不同染色体和连锁群,然后使用ggforce扩展包的geom_diagonal()函数添加标记在连锁图和物理图间对应位置的连线,最后用coord_polar()将直角坐标系转换为极坐标系。
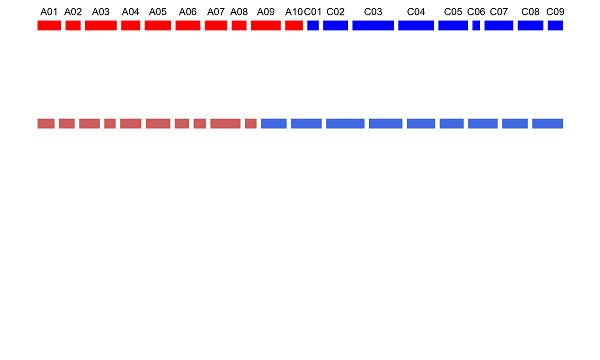
构建染色体和连锁群
每个染色体和连锁群长度不同,且都是从 0 开始,为了能在同一水平线上依次画出不同染色体或连锁群,我们需要把其起始位置 0 和终止位置加上该染色体或连锁群前面的长度总和,并且为了是不同染色体或连锁群不重合,还需要加上 n-1 (n为该染色体或连锁群排序位置)个间隔大小,同理标记在基因组或连锁图上的位置也需要相同处理。还有一个问题是物理图和遗传图总长以及单位并不相同,需要将长度信息和位置信息标准化,在此我们将所有 。
1 | |
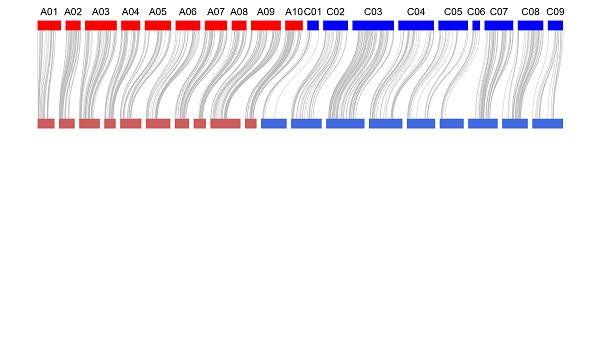
添加曲线
上述已经画出染色体和连锁群,我们用``函数添加一条bezier曲线用于表示标记在基因组和连锁图上对应的位置。
1 | |
坐标系变换
使用coord_polar()函数将直角坐标系变成极坐标系。
1 | |
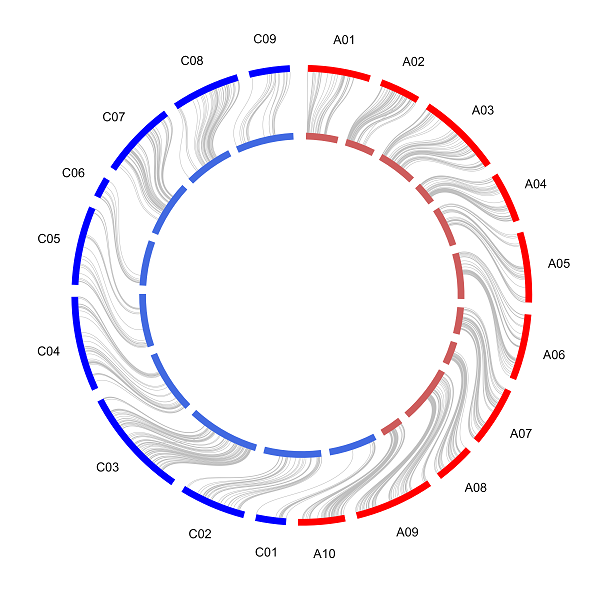
和文章中的circos图对比发现,我们纠正了不同连锁群间比例和不同染色体间比例的问题,使其按照各自的大小比例分布。但同时一个新的问题出现了,由于A亚基因组的在物理图中所占比例较低,在遗传图中所占比例略大,C亚基因组相反,也就是在物理图和遗传图中不同染色体和不同连锁群比例不同,导致了标记在物理图和遗传图间的位置偏差越来越大,表现在图中就是中间曲线被越拉越长。因此需要根据个人偏好选择两种不同形式的circos图。
图片来源
